Le coronavirus a-t-il été créé en laboratoire ?
Publié en ligne le 2 mai 2020 - Covid-19 -
Diverses rumeurs circulent depuis le début de l’épidémie de coronavirus selon lesquelles le virus serait une création humaine. Ces rumeurs se classent en deux catégories : celles qui avancent une hypothèse malveillante, et celles qui avancent une hypothèse accidentelle [1].
Dans le premier cas, le coronavirus serait une arme biologique délibérément créée et libérée dans le but de provoquer une épidémie ; dans le second, des chercheurs auraient joué aux apprentis-sorciers et auraient créé le virus dans un but peut-être légitime (à des fins de recherche), mais par laxisme ou incompétence, leur création leur aurait échappé et serait sortie du laboratoire. Entre les deux, on entend également l’hypothèse que le virus aurait été créé délibérément à des fins malveillantes (en tant qu’arme biologique) mais libéré de manière accidentelle – c’est ce qu’a avancé par exemple (sans aucune preuve) le Washington Times du 26 janvier [2], citant un analyste israélien pour qui le coronavirus est « lié au programme d’armement biologique chinois ».
Alors, qu’en est-il ?
Diversité des coronavirus
On parle couramment, depuis le début de l’épidémie, du coronavirus. Il s’agit néanmoins d’un abus de langage qui, s’il est aisément compréhensible, peut rapidement entraîner une confusion néfaste. Le virus causant l’épidémie actuelle est un coronavirus. Les coronavirus sont une famille de virus connue depuis longtemps puisque les premiers coronavirus pathogènes pour l’Homme ont été découverts dans les années 1960 [3]. Jusqu’en 2019, on connaissait six différents coronavirus affectant l’humain :
• quatre coronavirus endémiques (présents dans la population de manière habituelle) et faiblement pathogènes (CoV229E, CoV-OC43, CoV-NL63, et CoV-HKU1) qui, la plupart du temps, ne causent rien de plus grave qu’un rhume (ils sont collectivement la deuxième cause la plus fréquente des rhumes juste derrière les rhinovirus – on estime qu’environ un tiers des rhumes sont causés par ces quatre coronavirus) ;
• deux coronavirus hautement pathogènes, le SARS-CoV, responsable de l’épidémie de syndrome respiratoire aigu sévère (Sras, ou SARS en anglais pour Severe Acute Respiratory Syndrome) qui a fait 774 morts (principalement en Asie) en 2002-2003, et le MERS-CoV, responsable du syndrome respiratoire du Moyen-Orient (MERS, Middle East Respiratory Syndrome), qui depuis son émergence en 2012 a fait environ 750 morts.
Le virus responsable de l’épidémie actuelle est un nouveau coronavirus, qui vient s’ajouter aux six précédemment connus pour être pathogènes pour l’Homme. Il a d’ailleurs été initialement appelé nCoV2019, pour Novel Coronavirus 2019, avant d’être finalement baptisé SARS-CoV-2 en raison de sa ressemblance avec le coronavirus SARS-CoV de 2002-2003 [4].

Ces éclaircissements préliminaires permettent déjà d’établir que la mention « coronavirus » qui apparaît sur les étiquettes de produits désinfectants manufacturés bien avant 2019 (figure 1) n’est pas le signe qu’on vous cache quelque chose ou que les fabricants de désinfectants avaient anticipé l’épidémie avant tout le monde : cette mention fait seulement référence aux quatre coronavirus communs en partie responsables des rhumes, pas au SARS-CoV-2 responsable de l’épidémie actuelle.
Le brevet de l’Institut Pasteur
Une des théories qui circulent sur l’origine du SARS-CoV-2 est qu’il aurait été créé par l’Institut Pasteur. La preuve en serait un brevet déposé par des chercheurs dudit institut, qui porterait sur l’invention du virus. Le brevet en question existe bel et bien (figure 2) : c’est le brevet européen EP1694829B1 [5] (et les brevets équivalents déposés auprès des bureaux de brevets d’autres pays, comme le brevet 2007/0128224 aux États-Unis [6]).

Mais ce brevet :
• concerne le SARS-CoV (le coronavirus responsable de l’épidémie de Sras entre 2002 et 2003, comme mentionné ci-dessus), et non le SARS-CoV-2 ;
• décrit un virus découvert et non inventé, mais le jargon juridique employé, notamment pour les brevets, ne fait pas cette distinction (formellement tout ce qui est couvert par un brevet est une « invention »).
Plus précisément, le brevet porte sur la découverte d’une nouvelle souche de SARS-CoV, isolée à partir d’un échantillon prélevé sur un patient vietnamien souffrant du Sras – la souche est « nouvelle » parce qu’elle est très légèrement différente des autres souches de SARS-CoV précédemment découvertes au cours de l’épidémie de 2002-2003. Le brevet porte aussi sur la caractérisation de la souche (la détermination de son génome complet), sur la construction d’outils de biologie moléculaire permettant d’étudier le virus en laboratoire, et sur l’élaboration d’un prototype de vaccin.
Ce que prouve ce brevet, c’est qu’après une épidémie provoquée par un virus que l’on n’avait encore jamais vu, les chercheurs de l’Institut Pasteur se sont attelés à étudier ce virus et à mettre au point un vaccin au cas où on le verrait resurgir plus tard. Autrement dit, l’Institut Pasteur a fait exactement ce pourquoi il existe.
Le laboratoire P4 de Wuhan
On s’est étonné, dès le début de l’épidémie actuelle, de la présence d’un institut de recherche en virologie dans la ville de Wuhan, épicentre de l’épidémie – d’autant plus que cet institut abrite en son sein le seul laboratoire P4 de Chine, le Wuhan National Biosafety Laboratory (les laboratoires dits « P4 » sont des laboratoires de très haute sécurité spécialement conçus pour l’étude des agents pathogènes les plus dangereux). On a ainsi rapidement insinué que ce laboratoire serait d’une manière ou d’une autre à l’origine de l’épidémie.
Nous verrons plus loin que rien ne vient étayer cette hypothèse, mais arrêtons-nous d’abord un instant sur ce que certains présentent comme une « troublante coïncidence ».
Wuhan est une ville de plus de huit millions d’habitants, la septième ville la plus peuplée de Chine [7]. C’est la capitale de la région de Hubei et un centre économique et culturel majeur. C’est aussi et surtout un important pôle universitaire : la ville abrite des dizaines d’établissements d’enseignement supérieur et des dizaines d’organismes de recherche dans toutes les disciplines scientifiques. S’étonner de la présence d’un institut de virologie dans une grande ville universitaire comme Wuhan serait comme s’étonner de la présence de l’Institut Pasteur à Paris ou du laboratoire Jean Mérieux à Lyon.
Les épidémies tendent à éclater (ou à être détectées) dans les zones densément peuplées, comme les grandes métropoles (ainsi l’épidémie de Sras en 2002 à Foshan – 7 millions d’habitants –, la grippe pandémique de 1968 à Hong-Kong, la grippe pandémique de 1957 à Singapour...), et les grandes métropoles ont tendance à accueillir des organismes de recherche.
Comment sait-on que le virus n’est pas artificiel ?
Le brevet de l’Institut Pasteur susmentionné ne prouve pas que ledit institut a créé le virus, et la simple présence de l’Institut de virologie de Wuhan dans la région où l’épidémie a démarré ne suffit pas à prouver qu’il en est à l’origine. Mais quelles preuves a-t-on que le virus est d’origine naturelle ?
Un génome sans trace de manipulation
Les meilleures preuves nous sont fournies par le virus lui-même, et plus particulièrement son génome, dont on trouve une copie dans chaque particule virale. Le génome du SARS-CoV-2 a été rapidement séquencé et mis à disposition des chercheurs du monde entier[Pas seulement des chercheurs, en fait : les séquences sont disponibles pour quiconque les veut, dans GenBank par exemple.]. En l’étudiant, on peut apprendre un certain nombre de choses sur les origines probables ou improbables du virus.
Notamment, on peut noter d’emblée que l’on ne trouve dans le génome aucune trace des techniques de biologie moléculaire connues et utilisées en laboratoire pour manipuler ce genre de virus[Du fait de leur grande taille (≥ 30 000 bases, alors que la plupart des virus à ARN ont un génome n’excédant pas 10 000 bases), manipuler les génomes de coronavirus nécessite le développement d’outils de génétique inverse (comme des plasmides ou des chromosomes artificiels de levure) spécifiquement adaptés, et dont des fragments devraient être présents dans le génome du SARS-CoV-2 s’il était issu d’un de ces systèmes. ] [8]. Ce fait à lui seul rend hautement improbable que le virus soit issu d’une manipulation génétique, sauf à supposer que les manipulateurs ont utilisé des méthodes totalement inconnues du monde académique.
Portrait de famille du virus
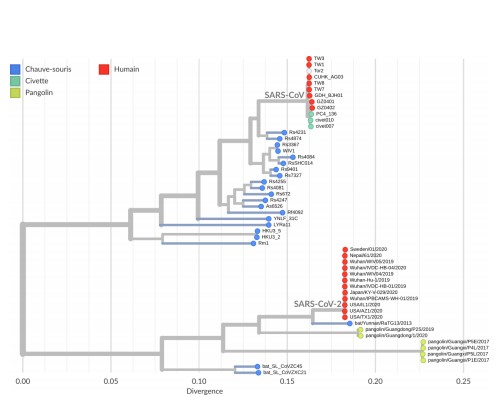
Ensuite, en comparant le génome du SARS-CoV-2 avec les génomes des coronavirus déjà connus (y compris les coronavirus animaux), on peut retracer « l’historique » de ce virus et en déduire son origine probable. C’est ce qu’on appelle une étude phylogénétique. C’est avec ce genre d’études qu’on a réalisé que le SARS-CoV-2 était apparenté au SARS-CoV de 2002-2003, avec lequel il partage environ 70 % de son génome [4] – c’est d’ailleurs la raison pour laquelle il a été baptisé ainsi.
Il fait donc partie de la famille grandissante des virus dits SARS-CoV-like, qui regroupe les virus apparentés au SARS-CoV (figure 3) : le SARS-CoV lui-même, et tous les virus similaires qui ont été identifiés depuis 2003 chez plusieurs espèces (le MERS-CoV de 2012 n’en fait pas partie : il s’agit certes d’un coronavirus, mais il est suffisamment différent pour appartenir à une famille distincte). Dans cette famille, le plus proche cousin du SARS-CoV-2 est un virus de chauve-souris appelé RaTG13, qui partage environ 96 % de son génome avec le SARS-CoV-2.
Bien que l’on ne puisse pas encore déterminer avec certitude la provenance du SARS-CoV-2, l’hypothèse de loin la plus vraisemblable à ce stade est qu’il dérive d’un virus de chauve-souris qui, de mutation en mutation, est devenu capable d’infecter des cellules humaines.
Des indices supplémentaires sur l’origine du virus
Une analyse plus détaillée du génome du SARS-CoV-2 révèle qu’il contient trois éléments caractéristiques qui le distinguent de ses cousins de la famille SARS-CoV-like [8] et qui nous fournissent des indices supplémentaires sur les origines du virus. Tous trois sont situés dans la partie du génome codant pour la protéine virale dite « S » sur laquelle il est nécessaire de s’attarder. La protéine S, ou spike protein, est située à la surface de la particule virale. Elle donne aux coronavirus leur apparence particulière qui leur a valu leur nom de « virus à couronne ».
La protéine S du SARS-CoV-2, comme celle de son cousin le SARS-CoV, reconnaît une protéine particulière exprimée à la surface des cellules épithéliales pulmonaires (expliquant pourquoi ce virus cause principalement des symptômes respiratoires). La protéine S est capable de se lier très fortement à la surface des cellules épithéliales, de cliver et fusionner la membrane qui entoure le virus avec la membrane cellulaire, permettant ainsi au virus de pénétrer dans la cellule épithéliale et de l’infecter. Le mécanisme de reconnaissance et de fusion des membranes fait intervenir, d’une part, un domaine de liaison avec la membrane de la cellule à infecter et, d’autre part, un site de clivage composé d’une séquence de quatre acides aminés et d’une partie chimiquement complexe composée d’acides aminés et de sucres de structures spécifiques. Le domaine de liaison du SARS-CoV-2 diffère de celui du SARS-CoV. Il entraîne une liaison très forte avec la membrane de la cellule à infecter (ce qui contribue à sa pathogénicité), mais cette affinité n’est pas optimale et pourrait être améliorée par des mutations. Cela suggère fortement que ce virus n’est pas le fruit d’une manipulation génétique visant à créer délibérément un coronavirus hautement pathogène. En effet, quiconque aurait voulu améliorer cette affinité aurait introduit ces mutations bien décrites dans la littérature scientifique. Quant au site de clivage, il comprend une séquence d’acides aminés et de sucres spécifiques qui suggère un contact avec un système immunitaire, ce qui rend peu probable que cette machinerie biochimique du SARS-CoV-2 soit issue d’une culture in vitro (voir les détails scientifiques en encadré).
Ce que nous apprend la protéine « S »
Une analyse détaillée du génome du SARS-CoV-2 révèle qu’il contient trois éléments caractéristiques qui le distinguent de ses cousins de la famille SARS-CoV-like [8] et qui nous fournissent des indices supplémentaires sur les origines du virus. Tous trois sont situés dans la partie du génome codant pour la protéine virale dite « S ».
La protéine S, ou spike protein, est située à la surface de la particule virale. Elle donne aux coronavirus cette apparence particulière qui leur a valu leur nom de « virus à couronne » (figure 4a). Elle est composée de deux domaines, S1 et S2 (figure 4b), chacun responsable d’une fonction essentielle au cycle de vie du virus : le domaine S1 reconnaît une protéine à la surface des cellules de l’hôte, permettant au virus de s’y accrocher ; le domaine S2 déclenche la fusion de l’enveloppe membranaire virale avec la membrane cellulaire, permettant au virus de pénétrer dans la cellule [10].
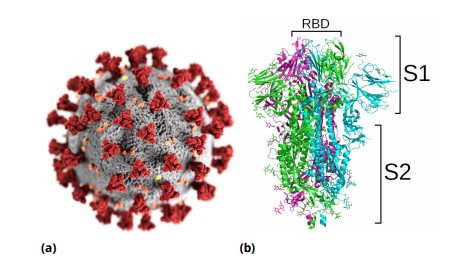
La protéine S du SARS-CoV-2, comme celle de son cousin le SARS-CoV, reconnaît une protéine appelée « enzyme 2 de conversion de l’angiotensine » (ACE2 pour Angiotensin-Converting Enzyme 2), notamment exprimée à la surface des cellules épithéliales pulmonaires (expliquant pourquoi ce virus cause principalement des symptômes respiratoires). L’interaction entre la protéine S et l’ACE2 dépend d’une petite partie précise du domaine S1 appelée le « domaine de liaison au récepteur » (RBD pour Receptor-Binding Domain). Une fois la protéine S liée à l’ACE2 à la surface d’une cellule pulmonaire, le domaine S1 est clivé afin de laisser le champ libre au domaine S2 pour fusionner la membrane virale avec la membrane cellulaire.
Les trois éléments qui distinguent la protéine S du SARS-CoV-2 sont : un RBD avec une forte affinité pour ACE2 ; la présence d’un site de clivage polybasique à la jonction entre les domaines S1 et S2 ; et la présence de sites de glycosylation au voisinage de cette jonction.
Le RBD du SARS-CoV-2 Le domaine de liaison à l’ACE2 du SARS-CoV2 diffère considérablement de celui des autres coronavirus apparentés au SARS-CoV – même le RaTG13, le coronavirus de chauve-souris dont on a vu qu’il était le plus proche cousin du SARS-CoV-2, possède un RBD sensiblement éloigné. Le RBD du SARS-CoV-2 est capable de se lier très fortement à l’ACE2, ce qui contribue certainement à la pathogénicité du virus. Néanmoins, il n’est pas optimal. De nombreuses études ont été réalisées au cours des quinze dernières années sur le RBD du SARS-CoV ; elles ont notamment permis de déterminer les mutations qui conféreraient à un coronavirus une affinité maximale pour l’ACE2 [12]. Toutefois on ne retrouve aucune de ces mutations dans le SARS-CoV-2. Cela suggère fortement que ce virus n’est pas le fruit d’une manipulation génétique visant à créer délibérément un coronavirus hautement pathogène : quiconque aurait voulu faire cela aurait vraisemblablement introduit ces mutations déjà connues pour rendre le virus plus efficace (elles ne sont pas secrètes, elles figurent dans la littérature scientifique !) au lieu d’inventer un RBD sous-optimal.
Le site de clivage polybasique Le SARS-Cov-2 contient une insertion de quatre acides aminés à la jonction entre les domaines S1 et S2 de la protéine S. Cette insertion entraîne la formation d’un site dit « polybasique »[Site polybasique : une courte séquence d’acides aminés dont la plupart sont chargés positivement.] qui est reconnu par une famille de protéases[Protéase : une enzyme dont l’activité consiste à couper une protéine.] naturellement présentes dans les cellules, comme la furine [15]. La protéine S pourrait ainsi être partiellement coupée dès la fabrication de la particule virale, et non pas seulement après la liaison à l’ACE2 ; cela faciliterait ainsi grandement l’entrée du virus dans ses cellules cibles. Rien ne suggère que cette insertion soit d’origine artificielle, elle peut parfaitement s’expliquer par l’évolution naturelle du virus. On sait en effet que ce genre de sites peut être acquis spontanément par mutation successive : la région au voisinage de la jonction entre les domaines S1 et S2 de la protéine S est connue pour muter facilement, et un site similaire avait d’ailleurs été trouvé dans le MERS-CoV de 2012. Des travaux réalisés sur des virus grippaux ont montré qu’on pouvait aussi voir évoluer ce genre de sites en cultivant un virus in vitro [16], mais aucun travail similaire n’a encore été décrit sur les coronavirus.
Récemment, un RBD comparable à celui du SARS-CoV-2 a été trouvé dans des coronavirus de pangolin [13]. La ressemblance est cependant limitée à ce domaine particulier et les virus de pangolin restent, sur l’ensemble de leur génome, plus éloignés du SARS-CoV-2 que le RaTG13 des chauves-souris. C’est sur ces observations que repose l’hypothèse récemment émise selon laquelle les pangolins ont pu servir d’intermédiaires lors de l’adaptation d’un virus de chauves-souris aux humains, mais cela reste à démontrer [14].
Les sites de glycosylation De part et d’autre du site de clivage polybasique, à la jonction entre les domaines S1 et S2, la protéine S contient trois sites de glycosylation, c’est-à-dire des acides aminés sur lesquels peuvent être greffés des sucres. La glycosylation des protéines virales est un phénomène bien connu chez d’autres virus, chez lesquels on pense qu’elle fournit une protection contre les défenses immunitaires de l’hôte [17], mais qui n’avait pas été observé à cet endroit jusqu’à présent dans les coronavirus apparentés au SARS-CoV. Comme les sites de glycosylation évoluent typiquement en réaction à la présence d’un système immunitaire (qui fait peser une pression de sélection sur le génome viral), leur présence dans la protéine S du SARS-CoV-2 rend peu probable que le virus soit issu d’une culture in vitro.
Récapitulatif
L’étude du génome du SARS-CoV-2 nous apprend donc que :
• l’hypothèse d’une création par manipulation génétique délibérée est hautement improbable de par (a) l’absence de toute trace des méthodes connues pour manipuler les coronavirus ; (b) le RBD non optimal, alors que les mutations qui rendraient le RBD optimal (et donc le virus encore plus infectieux) sont parfaitement connues depuis plusieurs années ;
• l’hypothèse d’une création accidentelle, par culture in vitro, est improbable de par (a) l’absence d’un coronavirus connu suffisamment proche pour que le SARS-CoV-2 puisse en être dérivé (le RaTG13, le plus proche cousin du SARS-CoV-2, en est déjà trop éloigné, surtout dans la région critique du RBD) ; (b) la présence des sites de glycosylation, qui suggère un contact avec un système immunitaire impossible à simuler in vitro ;
• l’hypothèse d’une évolution naturelle depuis un virus de chauves-souris ou de pangolin est de loin l’hypothèse la plus vraisemblable.
Merci à @LuciusLeVirus et @SciTania pour leur lecture critique du manuscrit.
1 | Franceinfo, « Coronavirus : pour plus d’un Français sur quatre, le Covid-19 a été créé en laboratoire, selon une étude », 28 mars 2020 (visité le 28/03/2020).
2 | Gertz B, “Coronavirus Link to China Biowarfare Program Possible, Analyst Says”, The Washington Times, 26 janvier 2020 (visité le 27/03/2020).
3 | Weston S, Frieman MB, “Respiratory Viruses”, in : Schmidt TM (dir.), Encyclopedia of Microbiology (Fourth Edition), Academic Press, 2019, 85-101 (visité le 21/03/2020).
4 | Gorbalenya AE et al., “The Species Severe Acute Respiratory Syndrome-Related Coronavirus : Classifying 2019-nCoV and Naming It SARS-CoV-2”, Nat Microbiol, 2020, 5:536-44 (visité le 27/03/2020).
5 | Kunst F et al., “Nouvelle souche de coronavirus associée au Sras et ses applications”, brevet européen 1694829B1, CNRS, Institut Pasteur, université Paris Diderot, 4 août 2010 (visité le 28/03/2020).
6 | van der Werf S et al., “Novel Strain of SARS-Associated Coronavirus and Applications Thereof”, Brevet américain 20070128224A1, CNRS, Institut Pasteur, université Paris Diderot, 7 juin 2007 (visité le 27/03/2020).
7 | “Wuhan : The London-Sized City Where the Virus Began”, BBC News, China, 23 janvier 2020 (visité le 27/03/2020).
8 | Andersen KG et al., “The Proximal Origin of SARS-CoV-2”, Nat Med, 2020, 26:450-2
9 | Hadfield J et al., “Nextstrain : Real-Time Tracking of Pathogen Evolution”, Bioinformatics, 2018, 34:4121-23 (visité le 27/03/2020).
10 | Tortorici MA, Veesler D, “Chapter Four – Structural Insights into Coronavirus Entry”, in : Rey FA (dir.), Advances in Virus Research, T. 105, “Complementary Strategies to Understand Virus Structure and Function”, Academic Press, 2019, 93-116 (visité le 28/03/2020).
11 | Walls AC et al. “Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein”, Cell, 2020, 181:281-92 (visité le 26/03/2020).
12 | Sheahan T et al., “Mechanisms of Zoonotic Severe Acute Respiratory Syndrome Coronavirus Host Range Expansion in Human Airway Epithelium”, J Virol, 2008, 82:2274-85.
13 | Lam TTY et al., “Identifying SARS-CoV-2 Related Coronaviruses in Malayan Pangolins”, Nature, 2020, doi:10.1038/s41586-020-2169-0 (visité le 30/03/2020).
14 | Wong MC et al., “Evidence of Recombination in Coronaviruses Implicating Pangolin Origins of nCoV-2019”, bioRxiv, 2020, doi:10.1101/2020.02.07.939207 (visité le 30/03/2020).
15 | Coutard B et al., “The Spike Glycoprotein of the New Coronavirus 2019-nCoV Contains a Furin-like Cleavage Site Absent in CoV of the Same Clade”, Antiviral Research, 2020, 176:104742 (visité le 28/03/2020).
16 | Ito T et al., “Generation of a Highly Pathogenic Avian Influenza A Virus from an Avirulent Field Isolate by Passaging in Chickens”, J Virol, 2011, 75:4439-43.
17 | Bagdonaite I, Wandall HH, “Global Aspects of Viral Glycosylation”, Glycobiology, 2018, 28:443-67 (visité le 25/03/2020).
Partager cet article
L'auteur
Damien Goutte-Gattat

Damien Goutte-Gattat est chercheur post-doctorant au Barts Cancer Institute, Queen Mary University of London (2020).
Plus d'informationsCovid-19

Covid-19, hydroxychloroquine et traitement médiatique
Le 9 janvier 2023
Mauvaises conduites et Covid-19
Le 11 octobre 2022
Restaurer l’intégrité scientifique après la crise Covid-19
Le 4 octobre 2022Communiqués de l'AFIS
















![[16 juin 2022 - Paris] Covid-19 : Deux ans d'épidémie, qu'avons-nous appris ?](local/cache-gd2/0f/33c69087dceb597afeb0e562895a7f.jpg?1724967507)








